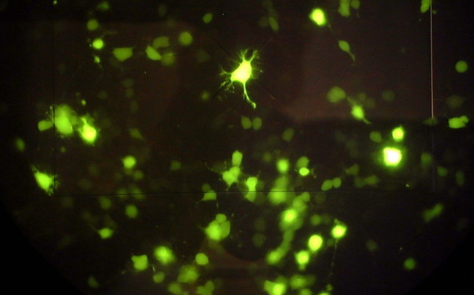
EndoTrack
Chaînes de Markov cachées hybrides pour la cartographie du trafic intracellulaires dans les tissus 3D en développement.
Molécules de type Rab5 (en vert) et Dextran (en mauve) imagées par spinning-disk confocal © Claudio Collinet (IBDM)
Axe Vivant
Mathématiques
∩ Biologie
Aperçu
Cartographier le trafic intracellulaire en tissus 3D par la modélisation statistique du mouvement et l’apprentissage à grande échelle.

Mots clefs
Chaînes de markov cachées, détection et suivi de particules multiples, apprentissage automatique, architecture transformer, morphogenèse tissulaire, dynamiques collectives multi-échelles, trafic vésiculaire intracellulaire, adhésion intercellulaire.
Coordination
- Philippe Roudot (Mathématiques pour le traitement du signal, Institut Fresnel, UMR7249, Aix-Marseille Université, Centrale Méditerranée, CNRS)
- Claudio Collinet (Biologie du développement, IBDM, Aix-Marseille Université, CNRS)
Financement
- 419 945 € sur 48 mois (Projet scientifique lauréat de l’appel à projets 2024)
Résumé
Le projet scientifique EndoTrack s’attaque à un verrou majeur de la compréhension du vivant : cartographier les dynamiques et les flux de transport intracellulaires au sein de cellules pendant la morphogenèse des tissus, c’est-à-dire au moment où se construit l’architecture fonctionnelle des organismes. Mesurer ces dynamiques est essentiel pour comprendre l’émergence des formes biologiques, mais aussi les dérégulations du développement à l’origine de pathologies. Or, dans ces environnements vivants complexes, la densité cellulaire, les mouvements tissulaires et la diffusion de la lumière rendent inopérantes les approches actuelles de suivi de particules.
Pour dépasser ce verrou, EndoTrack développe une approche hybride combinant chaînes de Markov cachées et architectures transformer, conçue pour estimer simultanément l’état latent des particules et la matrice d’association la plus probable à grande échelle. Cette synergie crée un cadre unifié où la puissance du deep learning est couplée aux garanties statistiques du filtrage bayésien. En parallèle, EndoTrack intègre ce développement algorithmique à une conception expérimentale en microscopie reposant sur un échantillonnage et une illumination optimisée pour maximiser l’information dynamique tout en limitant le bruit et la phototoxicité.
Cette méthodologie ouvre la voie à la première cartographie dynamique du trafic intracellulaire à l’échelle tissulaire dans un embryon en développement, avec une résolution spatio-temporelle suffisante pour quantifier les flux moléculaires au cœur de la morphogenèse. Au-delà de ce cas d’étude, EndoTrack établit un cadre générique pour l’analyse des dynamiques collectives dans les systèmes complexes, offrant des perspectives en biologie, physique statistique et intelligence artificielle.
- Garantir la scalabilité des algorithmes jusqu’à la densité des processus tissulaire à l’échelle sub-cellulaire.
- Unifier apprentissage automatique et filtrage bayésien dans un cadre interprétable.
- Modéliser des dynamiques intracellulaires avec un faible à priori sur les trajectoires.
- Outils mathématiques : Chaînes de Markov cachées, filtrage bayésien, modèles à attention (transformer), optimisation combinatoire, analyse stochastique.
- Données : Données de microscopie 4D (3D+t), imagerie par illumination aléatoire profonde, trajectoires simulées in silico, enregistrements in vivo dans l’embryon de drosophile.
- Comprendre les mécanismes fondamentaux du développement embryonnaire et de leurs dérégulations pathologiques.
- Réduire le coût énergétique de l’IA grâce à des approches hybrides parcimonieuses basées sur la modélisation statistique.
- Limiter les hallucinations liées à l’IA en ancrant l’apprentissage dans un cadre statistique contrôlé et interprétable.
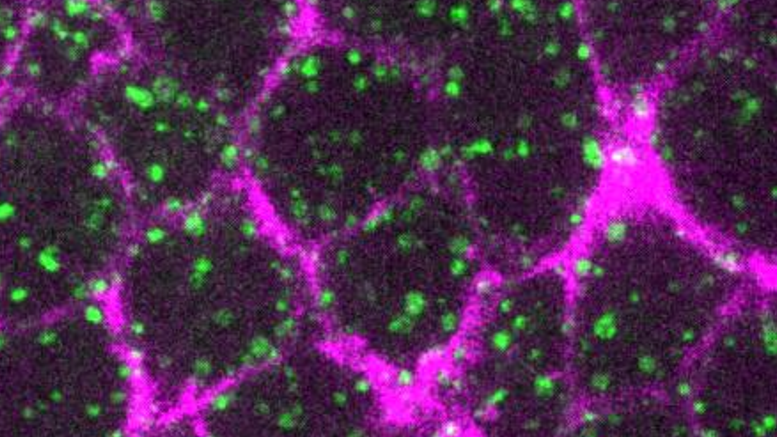
© Claudio Collinet (IBDM)

© Claudio Collinet (IBDM)
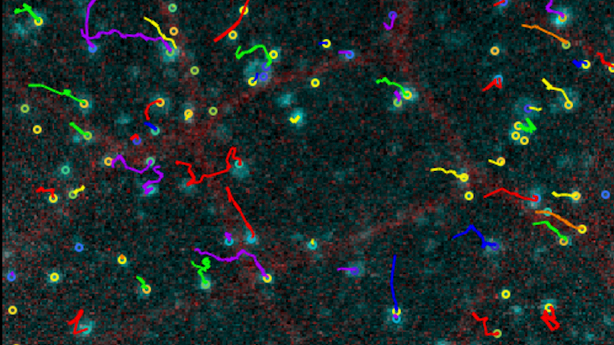
© Image acquise par Claudio Collinet (IBDM), traitement effectué par Philippe Roudot (Fresnel)
Recrutements envisagés
- 2 doctorats
Plus de projets